在基因组遗传变异的研究方面,有一个方式叫做单核苷酸多态性。
单核苷酸多态性(single nucleotide polymorphism,SNP)这一概念首次出现于1994年发表的人类分子遗传杂志上,随后于1996年由美国麻省理工的Lander正式提出。
SNP研究被认为是联系基因型和表现型之间关系的桥梁,是研究人类基因组计划走向应用的重要步骤。
什么是单核苷酸多态性?
想要了解SNP,就得先了解什么是DNA的多态性。
DNA的多态性指正常人群中,DNA分子或基因的某些位点可以发生中性改变,使DNA的结构各不相同,但并不影响基因的表达,形成多态。
DNA的多态性可以看作是在分子水平上的个体区别的遗传标志。
DNA的多态性可分为DNA片段长度多态性、DNA重复序列的多态性和单核苷酸多态性等。
■ DNA片段长度多态性是由于单个碱基的缺失、重复和插入所引起限制性内切酶位点的变化,导致DNA片段长度的变化。
■ DNA重复序列的多态性,主要表现于重复序列拷贝数的变异。
■ 而单核苷酸多态性(SNP)主要是指由单个核苷酸碱基(A/T/C/G)的变异(存在于1%以上的普通人群中)所引起的DNA序列多态性。
理论上讲,SNP既可能是二等位多态性,也可能是3个或4个等位多态性,但实际上,后两者非常少见,几乎可以忽略。因此,通常所说的SNP都是二等位多态性的。
这种变异可能是转换,也可能是颠换。
■ 转换(transition):它涉及嘌呤(即,腺嘌呤[A]和鸟嘌呤[G])与另一种嘌呤之间的替换,或嘧啶(即,胸腺嘧啶[T]和胞嘧啶[C])与另一种嘧啶之间的替换。例如:A↔G 或 C↔T
■ 颠换(transversion):它涉及一个嘌呤与一个嘧啶之间的替换。例如:A↔C、A↔T、G↔C 或 G↔T
通常情况下,SNP多为转换,即一种嘧啶碱基换为另一种嘧啶碱基,或一种嘌呤碱基换为另一种嘌呤碱基,转换与颠换之比为2:1。
研究证明,转换的几率之所以高,可能是因为CpG二核苷酸上的胞嘧啶残基是人类基因组中最易发生突变的位点,其中大多数是甲基化的,可自发地脱去氨基而形成胸腺嘧啶。
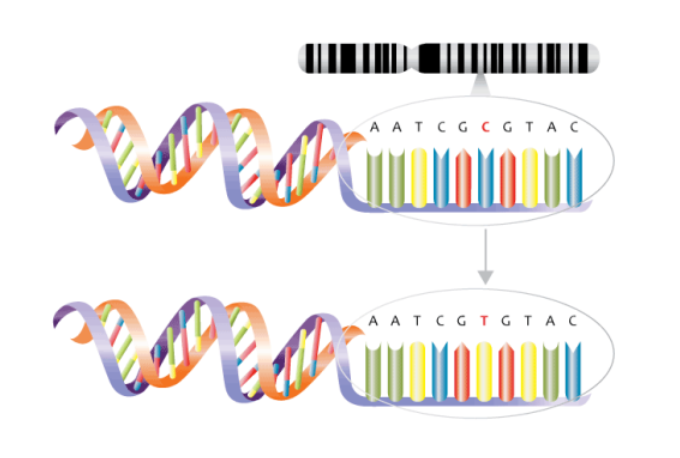
SNP在基因组内的形式有哪些?
在基因组DNA中,任何碱基均有可能发生变异,因此SNP既有可能在基因序列内,也有可能在基因以外的非编码序列上。
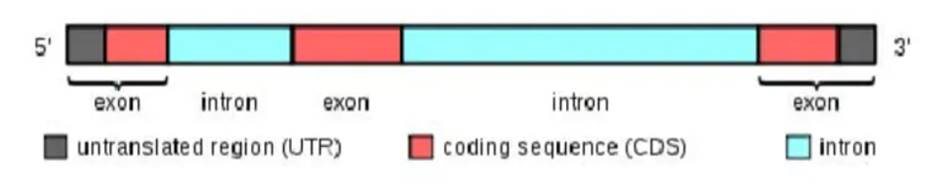
SNP分布不均匀,绝大多数位于蛋白的非编码区。位于非编码区域的SNP可细分为两类:
■ 内含子中SNP对基因功能的影响相对较小,主要依靠影响剪切位点活性来影响翻译。
■ 而基因调控区域包启动子区域、增强子区域等等,这些区域含有很多基因表达调控元件,这些位点的SNP发生变化,就会导致与调控因子的结合能力发生改变,从而影响基因表达。
位于编码区内的SNP比较少,但它在遗传性疾病研究中却具有重要意义,因此编码区内的SNP的研究更受关注。
位于编码区的SNP由于密码子的简并性原则的存在,它的发生不一定会引起蛋白质中氨基酸序列的改变,可分为同义SNP和非同义SNP两种类型。同义SNP并不影响其所翻译的蛋白质的氨基酸序列,而非同义SNP则会改变蛋白质的氨基酸序列,从而影响了蛋白质的功能,这种改变常是导致生物性状改变的直接原因。
SNP与人类健康
从以上介绍可以看出,有些SNP位于编码区域,可能导致氨基酸的改变或者影响基因的表达。有一些SNP位点已被证明在人类健康研究中非常重要。人体许多表型差异、对药物或疾病的易感性等等都可能与SNP有关。
根据与疾病的相关性,SNP分为中性SNP,疾病相关SNP和疾病易感性SNP。目前人们已经通过研究SNP图谱更深刻地认识到了癌症、糖尿病、某些神经性疾病等疾病的发生机制。
同时,研究人员发现,SNP可能有助于预测个体对某些药物的反应、对毒素等环境因素的易感性以及罹患特定疾病的风险。
概括来说,在临床和科研中,SNP多用于疾病的分子遗传机制研究、疾病基因定位、药物敏感或疾病易感性位点筛选等。
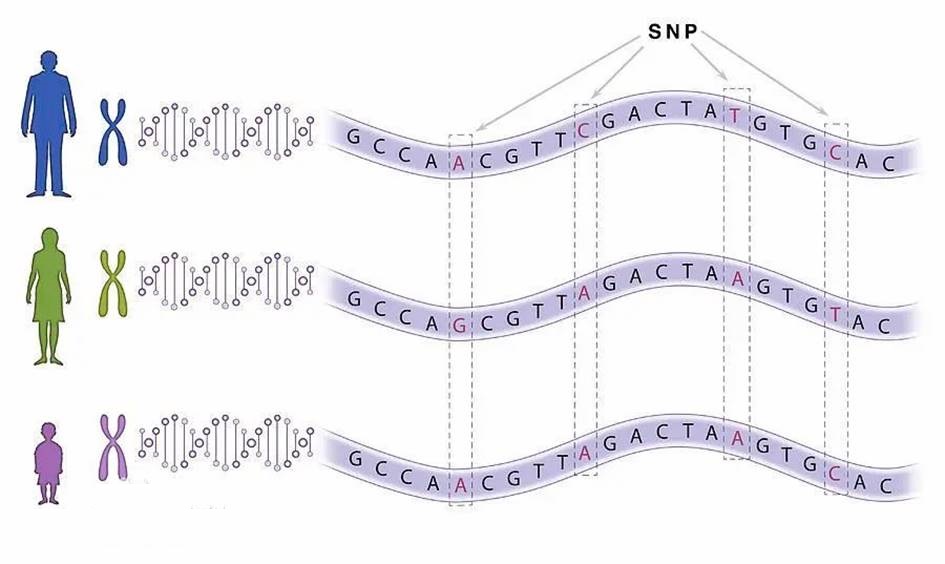
SNP的特点
SNP是人类可遗传的变异中最常见的一种,具有数量多,分布广,遗传稳定的特征。
■ 分布广、数量多:平均每500-1000个碱基对中就会出现1个SNP,这意味着一个人的基因组中大约有4-900万个SNP。科学家们在世界各地的人群中发现了超过6亿个SNP。
■ 富有代表性:某些位于基因内部的SNP有可能直接影响蛋白结构或者表达水平,它们可能为疾病遗传机理中的某些作用因素。
■ 易于分析:组成DNA的碱基虽然有4种,但SNP一般只有两种碱基组成,所以它是一种二态的标记。SNPs的二态性有利于其进行基因分型,而且可实现自动化。
另外,相对于其他的DNA多态性,SNP具有遗传稳定性,加上其检测易于自动化和批量化的特点,使得SNP在DNA多态性的研究中脱颖而出,被认为是新一代的遗传标记(继微卫星标记之后的第三代遗传标记)。
SNP自身的特性决定了它更适合于对复杂性状与疾病的遗传解剖以及基于群体的基因识别等方面的研究。SNP作为第三代分子标记,被广泛应用于分子遗传学、疾病诊治、分子育种和种群生态评估等诸多领域。
SNP的研究思路?
SNP的研究主要分为SNP的发现及SNP的基因分型。SNP的发现是应用的基础(通常是基于测序技术,利用已有数据库,对多个样本进行重测序发现的),而SNP的基因分型是应用的技术手段。一般按以下思路来设计实验:
(1)首先寻找目标SNP位点。
(2)然后,采用对照组和实验组的大量样本,对目标SNP位点进行验证。
(3)最后,对SNP分型结果与实验目的进行关联分析,如与疾病的关联分析、遗传连锁分析、品种鉴定等。
如何检测SNP?
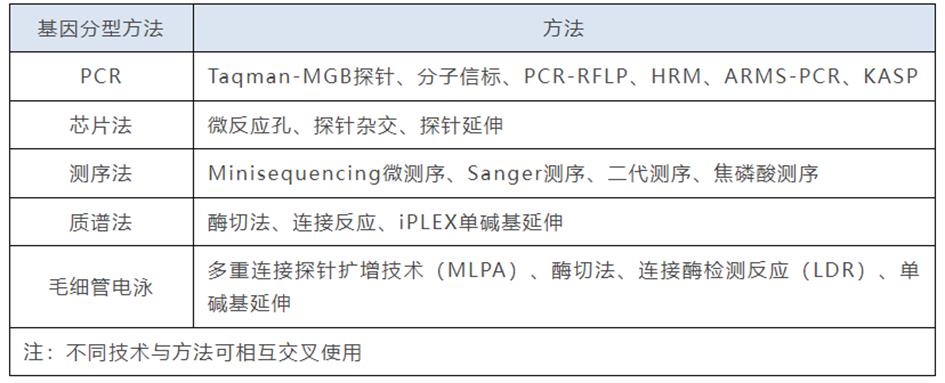
用于检测SNP的方法,除了测序法可直接获取核酸信息外,其它方法均是基于已知SNP位点进行设计的检测方法。
(1)测序法
Sanger测序是DNA序列分析的经典方法,可直接获取核酸序列信息,是SNP检测的“金标准”。
(2)TapMan探针法
在测试时,需筛选测试较多的探针,探针合成成本相对较高,因此TaqMan探针法通常用于少量SNP位点大量样本的分析,且对于近距离SNP位点探针设计也是比较困难的。
(3)ARMS-PCR法
ARMS-PCR方法检测灵敏度较高,且该方法在设计上需要的目的片段很短。缺点在于通量较低,无法在单次运行中检测多个SNP;不能检测未知突变;不适于位点附近GC含量过高或过低的SNP的分型检测。
(4)分子信标法
该方法与TaqMan探针法类似,均是通过探针中单个碱基的识别能力区分SNP。只是分子信标采用茎环结构,而TaqMan探针使用MGB修饰,以提高探针对SNP的识别特异性。
(5)高分辨率熔解曲法(HRM)
实验操作简单,不需要探针,成本相对较低,并且可以发现未知突变(但不能知道具体的突变类型)。但对实验仪器的温度分辨率要求相当高。
(6)CAPS法
CAPs法,又称限制性片段长度多态性聚合酶链反应(PCR-RFLP),是PCR技术与RFLP技术相结合的检测方法。CAPS仅能检测位于酶切位点处的SNP,对于酶切位点以外的SNP则无法检测。
(7)SNaPshot法
该技术优势是:分型准确(其准确度仅亚于直接测序),可多位点同时检测(最多可同时检测12个位点),可检测出受污染的样本。
(8)KASP法
KASP技术具有灵活、便宜、准确(检出率高达99.5%以上)等特点。
它是以常规PCR和荧光检测为基础,对设备要求较低,普通分子实验室完全能够满足开展实验的要求,具有良好的兼容性。
相比于其他技术,具有一定的灵活性,更容易实现高通量和自动化检测。另外,KASP采用的是通用探针,可以与各种不同的基因特异引物配合使用,而不需要针对每个特定的位点进行探针合成,这极大降低了实验的试剂成本。
来源:中洪博元














